Die meisten Biomoleküle funktionieren innerhalb der Zelle. Biochemische Tests und Analyseinstrumente werden jedoch hauptsächlich in vitro, in wässrigen Pufferlösungen oder Kristallen angewandt. Unser Forschungsziel ist die Entdeckung neuer biochemischer Funktionen durch die Untersuchung biomolekularer Reaktionen direkt in Zellen unter gesunden und kranken Bedingungen. Um biomolekulare Reaktionen in vivo mit hoher räumlicher und zeitlicher Auflösung zu analysieren, haben wir neuartige spektroskopische und mikroskopische Techniken in der Zelle entwickelt, die von der temperaturinduzierten Relaxation bis zur Einzelmolekülspektroskopie reichen. Wir wenden diese Methoden in immer komplexeren Umgebungen an, von zytomimetischen Medien und Bakterien bis hin zu (neuronalen) Zellen und multizellulären Organismen. Unser derzeitiges Forschungsinteresse liegt auf dem Gebiet der neurodegenerativen Erkrankungen mit Schwerpunkt auf den folgenden Forschungsthemen.
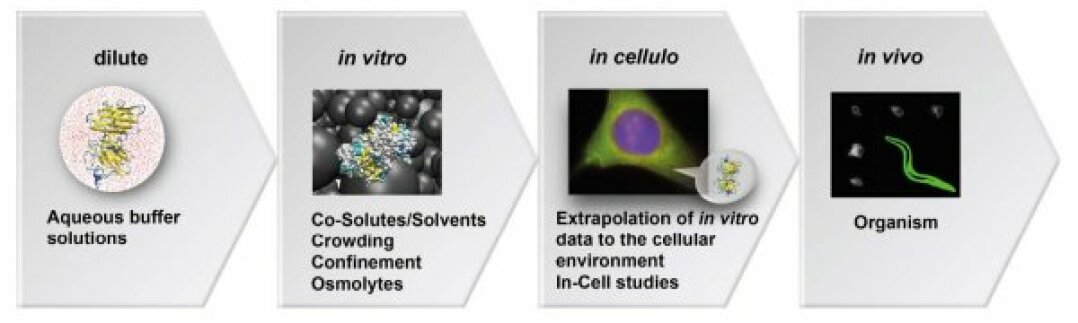
Die meisten Methoden zur Messung der Proteinfaltung und -aggregation von krankheitsrelevanten Proteinen beruhen auf In-vitro-Methoden wie ThT-Binging-Assays. Anhand von Experimenten in zytomimetischen Medien hat unser Labor ein neuartiges thermodynamisches Modell entwickelt, um zu verstehen, wie verschiedene Verbindungen wie Ionen[1], Stoffwechselprodukte[2,3], Verdränger[4], Medikamente[5] oder Dehydrierung[6] die zugrunde liegenden Gleichgewichte verändern können. Mithilfe eines Biosensor-Rahmens konnten wir Faltungsexperimente in der Zelle unter physiologischen Bedingungen[7-10], unter Zellstress[6,9,11,12] oder bei der Differenzierung[12] interpretieren. Diese Erkenntnisse führten zu einer neuen Klassifizierung krankheitsbedingter Mutanten[2,3,13,14]. Darüber hinaus haben wir einen neuartigen Assay zur Messung der Proteinaggregationskinetik mit hoher räumlich-zeitlicher Auflösung direkt in Zellen[5,13,15] entwickelt, der zum Screening neuer Klassen von Aggregationshemmern als potenzielle Medikamente[5,16] eingesetzt werden kann. In einem DFG-Projekt mit Prof. K. Huber (Paderborn) entschlüsseln wir nun die verschiedenen treibenden Kräfte, die die Aggregationskinetik in der Zelle bestimmen.
Molekulare Chaperone sind ein zellulärer Mechanismus zur Verhinderung von Aggregation und zur Aufrechterhaltung der räumlichen und zeitlichen Faltung des Proteoms. Im Rahmen eines HFSP-Projekts arbeiteten wir an der Entwicklung einer neuen Methode zur Messung der Proteostasekapazität unter verschiedenen zellulären Bedingungen[9,10]. Diese Arbeit veranlasste uns, die verschiedenen Mechanismen zu untersuchen, mit denen Chaperone die Faltung, Fehlfaltung und Aggregationsrate verschiedener Proteinsubstrate beeinflussen.
Es ist inzwischen klar, dass biomolekulare Kondensate eine wichtige Rolle bei der Faltung, Aggregation und Amyloid-assoziierten Krankheiten spielen. Unsere aktuelle Arbeit wird das Verständnis von funktionellen und dysfunktionellen Kondensaten voranbringen, indem wir aufzeigen, wie Stressgranula in Verbindung mit anderen Faktoren wie molekularen Chaperonen die Faltungsund Aggregationswege neu gestalten können, um die Proteinhomöostase zu unterstützen, oder umgekehrt, wie die Integrität von SGs durch die Sequestrierung verschiedener amyloidogener Spezies gefährdet werden kann. In der ersten Förderperiode des DFG-SPP2191 konnten wir zeigen, dass frühe Faltungsereignisse eine Rolle bei der Sequestrierung von Proteinen[14,17] spielen, wie z.B. bei pathogenen Mutanten des Enzyms SOD1[14]. In der zweiten Förderperiode werden wir unsere Studien ausweiten, um die Faltungsund Aggregationswege in den Stressgranula im direkten Vergleich zum Zytoplasma zu analysieren, was durch konfokales Fast Relaxation Imaging möglich sein wird, eine Methode, die kürzlich in unserem Labor entwickelt wurde.
Stoffwechselenzyme können in Zellen räumlich und zeitlich organisiert sein und regulatorische Multienzymkomplexe, so genannte Metabolone, bilden. Über solche Komplexe ist nur wenig bekannt, da sie nicht mit störenden Protokollen in vitro untersucht werden können. Auf der Grundlage unserer früheren Arbeiten, in denen wir gelernt haben, wie sich die Stabilität und Katalyse von Enzymen in zytomimetischen und zytoplasmatischen Umgebungen im Vergleich zu verdünnter Lösung verändert[3,18-20], untersuchen wir die Stabilität und metabolische Aktivität von Enzymen in Metabolons (GRK 2223: Zusammenbau von Proteinkomplexen).
Im Vergleich zu Proteinen ist über die Struktur und Faltung von RNA in Zellen nur wenig bekannt. Unser Labor war das erste, das die Stabilität gut gefalteter RNA-Hairpins in Zellen gemessen hat[8]. In unserer aktuellen Forschung konzentrieren wir uns auf krankheitsrelevante RNA-Sequenzen wie das CAG-Triplett-Repeat-RNA-Hairpin, das bei der Huntington-Krankheit eine Rolle spielt[21].
[1] Senske, M. et al. The temperature dependence of the Hofmeister series: thermodynamic fingerprints of cosolute–protein interactions. Phys. Chem. Chem. Phys. 18, 29698–29708 (2016).
[2] Gnutt, D. et al. Stability Effect of Quinary Interactions Reversed by Single Point Mutations. J. Am. Chem. Soc. 141, 4660–4669 (2019).
[3] Brylski, O. et al. Cellular ATP Levels Determine the Stability of a Nucleotide Kinase. Front. Mol. Biosci. 8, 1259 (2021).
[4] Gao, M. et al. Modulation of human IAPP fibrillation: cosolutes, crowders and chaperones. Phys. Chem. Chem. Phys. 17, 8338–8348 (2015).
[5] Vöpel, T. et al. Inhibition of Huntingtin Exon-1 Aggregation by the Molecular Tweezer CLR01. J. Am. Chem. Soc. 139, 5640–5643 (2017).
[6] Gnutt, D. et al. Imperfect crowding adaptation of mammalian cells towards osmotic stress and its modulation by osmolytes. Mol. BioSyst. 13, 2218–2221 (2017).
[7] Gnutt, D. et al. Excluded-Volume Effects in Living Cells. Angew. Chem. Int. Ed. 54, 2548–2551 (2015).
[8] Gao, M. et al. RNA Hairpin Folding in the Crowded Cell. Angew. Chem. Int. Ed. 55, 3224–3228 (2016).
[9] Wood, R. J. et al. A biosensor-based framework to measure latent proteostasis capacity. Nat Commun 9, 287 (2018).
[10] Raeburn, C. B. et al. A biosensor of protein foldedness identifies increased “holdase” activity of chaperones in the nucleus following increased cytosolic protein aggregation. J. Biol. Chem. 298, 102158 (2022).
[11] Hämisch, B. et al. Self‐Assembly of Pseudo‐Isocyanine Chloride as a Sensor for Macromolecular Crowding In Vitro and In Vivo. Chem. Eur. J. 26, 7041–7050 (2020).
[12] Gnutt, D. et al. Protein Folding Modulation in Cells Subject to Differentiation and Stress. Front. Mol. Biosci. 6, 38 (2019).
[13] Brylski, O. et al. Disease-Related Protein Variants of the Highly Conserved Enzyme PAPSS2 Show Marginal Stability and Aggregation in Cells. Front. Mol. Biosci. 9, (2022).
[14] Samanta, N. et al. Sequestration of Proteins in Stress Granules Relies on the In-Cell but Not the In Vitro Folding Stability. J. Am. Chem. Soc. 143, 19909–19918 (2021).
[15] Büning, S. et al. Conformational dynamics and self-association of intrinsically disordered Huntingtin exon 1 in cells. Phys. Chem. Chem. Phys. 19, 10738–10747 (2017).
[16] Azoulay-Ginsburg, S. et al. Chemical chaperones targeted to the endoplasmic reticulum (ER) and lysosome prevented neurodegeneration in a C9orf72 repeat expansion drosophila amyotrophic lateral sclerosis (ALS) model. Pharmacol. Rep (2021).
[17] Ribeiro, S. et al. Protein folding and quinary interactions: creating cellular organisation through functional disorder. FEBS Letters 592, 3040–3053 (2018).
[18] Dirkmann, M. et al. A Multiperspective Approach to Solvent Regulation of Enzymatic Activity: HMG-CoA Reductase. ChemBioChem 19, 153–158 (2018).
[19] Dhar, A. et al. Structure, function, and folding of phosphoglycerate kinase are strongly perturbed by macromolecular crowding. Proc. Natl. Acad. Sci. 107, 17586–17591 (2010).
[20] Ganguly, A. et al. Elucidation of the Catalytic Mechanism of a Miniature Zinc Finger Hydrolase. J. Phys. Chem. B 121, 6390–6398 (2017).
[21] Hautke, A. C. et al. S. Folding Stability and Self-Association of a Triplet-Repeat (CAG)20 RNA Hairpin in Cytomimetic Media. ChemSystemsChem 3, e2000052 (2021).
Hinweis: Beim Klick auf den Play-Button wird eine Verbindung mit einer RUB-externen Website hergestellt, die eventuell weniger strengen Datenschutzrichtlinien unterliegt und gegebenenfalls personenbezogene Daten erhebt. Weitere Informationen finden Sie in unserer Datenschutzerklärung.
Relaxationskinetik in lebenden Zellen
Proteomik enthüllt: Proteine organisieren sich zu Quinärstrukturen
Das Innere der Zelle: Ein komplexes Lösungsmittel
Understanding the 'Native' Solvent - from the Test Tube into the Cell